分子模拟技术栈
分子模拟技术栈
学习参考 .."LAMMPS data文件创建工具--moltemplate", Roy Kid
gaussview 绘制基本小分子,导出pdb文件
packmol 导入上一步的pdb文件,进行空间排布,导出系统的pdb文件
moltemplate 导入系统的pdb文件,同时提供力场信息,生成lammps模拟所需的data文件和力场文件
lammps 模拟
虽然moltemplate本身就具备建模功能,但是必须显式指定各原子和分子的位置,长链烷烃还比较好算,带支链的环烷烃等就比较复杂了.. 上面的一套流程不需要指定位置,直接建模就可以了.. 之后在moltemplate里给出力场信息.. 也就是说模型的构建和力场基本上是解耦的,这给MD尤其是在lammps中进行MD模拟提供了很大的方便,也可以较简单地切换不同力场进行比较.
622 decane +118 dodecane + 260 butylbenzene 均相体系为例
gaussview绘制基本单元
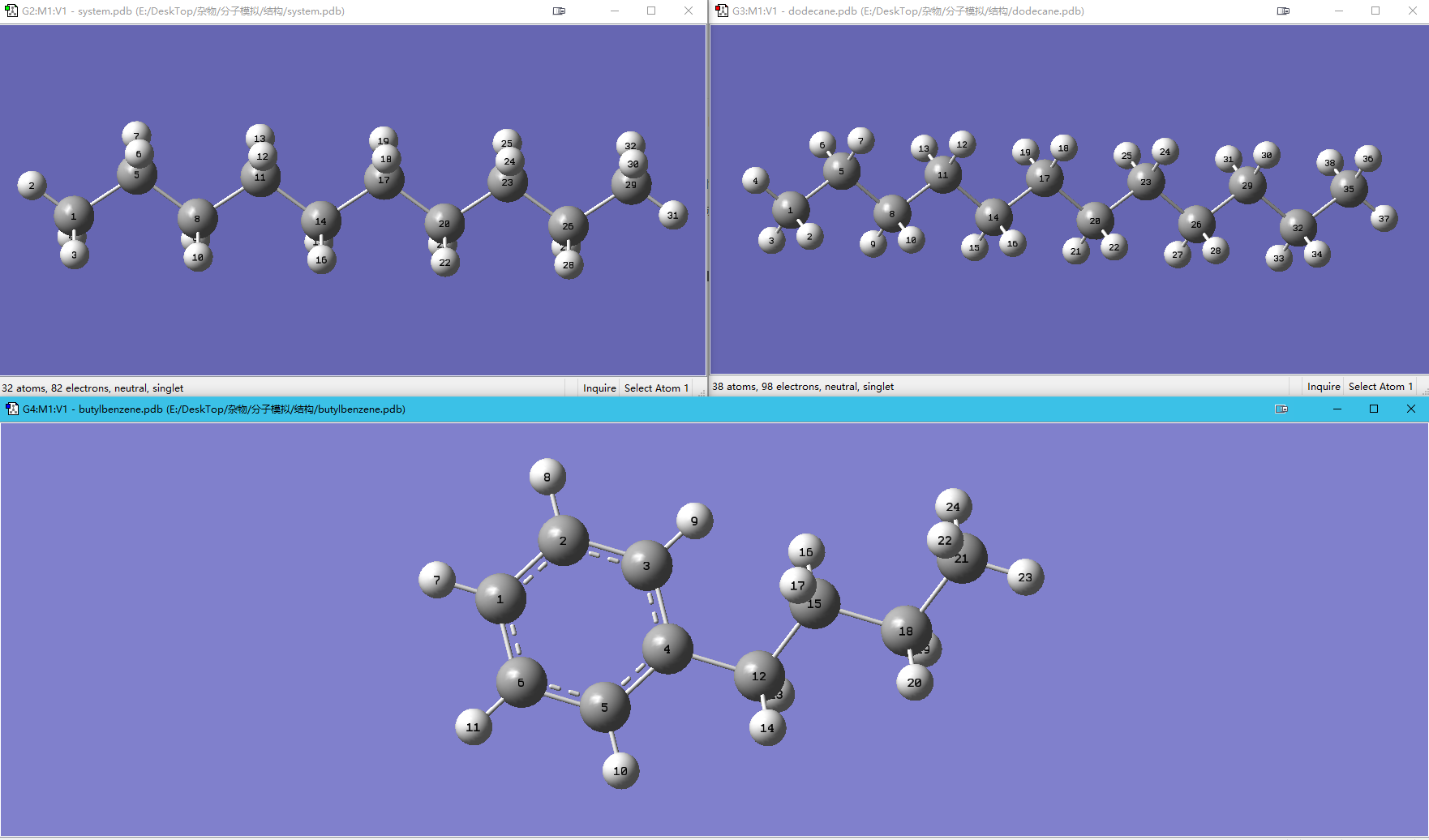
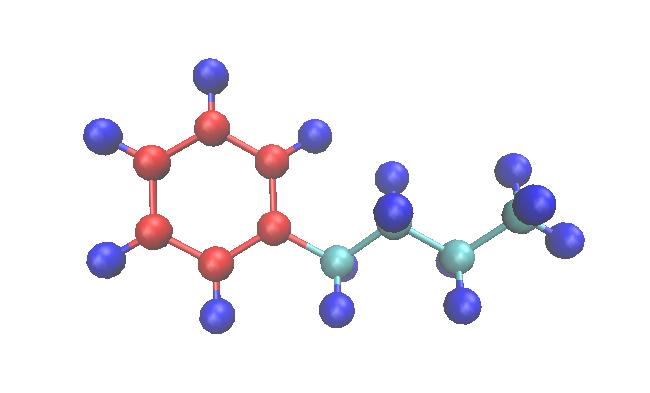
- 使用gaussview绘制出小分子,导出pdb文件,如下图所示:
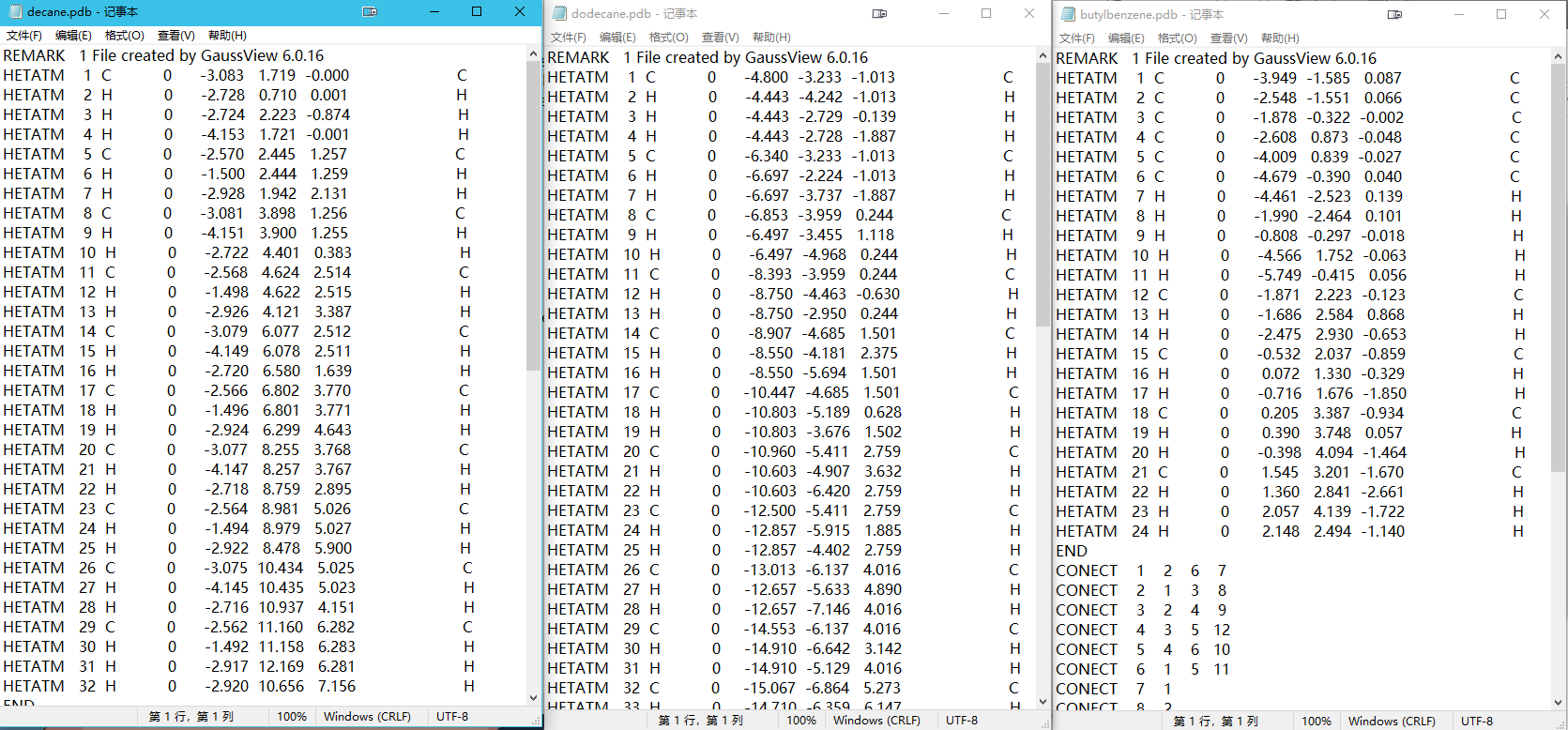
- 这里元素种类,名称等均不重要,唯一有用的就是各个原子的顺序,比如decane.pdb中定义原子的顺序和图中label的顺序是一样的,先是定义了CH3中的四个原子,然后是剩下链中的八个原子,最后是链尾端的CH3,每一个基团定义原子的顺序都是从C到H. Moltemplate中定义分子和系统信息时原子的顺序必须和pdb中的顺序完全一致,否则就会出现问题.
- 用packmol进行空间排布
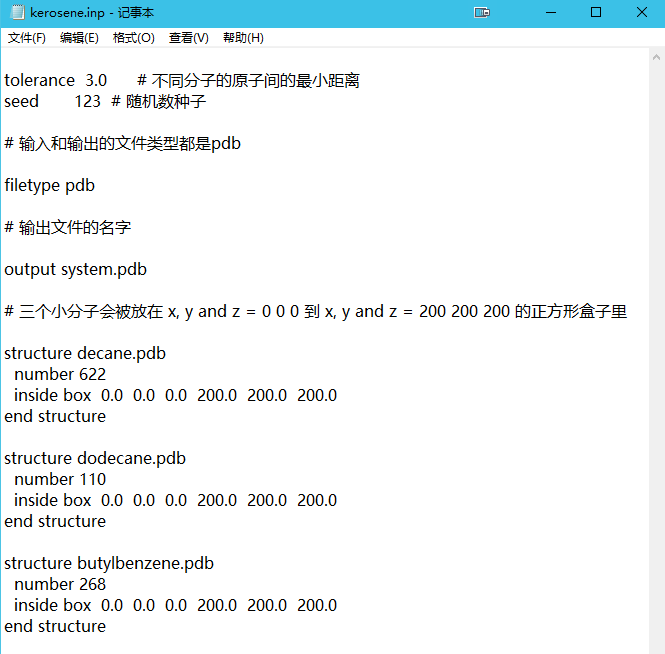
packmol的输入文件是.inp文件,使用时将packmol.exe放到工作文件夹里,通过cmd运行:
1
packmol < xxxx.inp
可以支持很复杂的结构定义,这里均相体系就很简单了. 需要指定tolerance,filetype,output,以及要填进去的东西
这一步会生成系统的结构文件,也就是.inp文件中指定的system.pdb
- 使用moltemplate给出力场信息
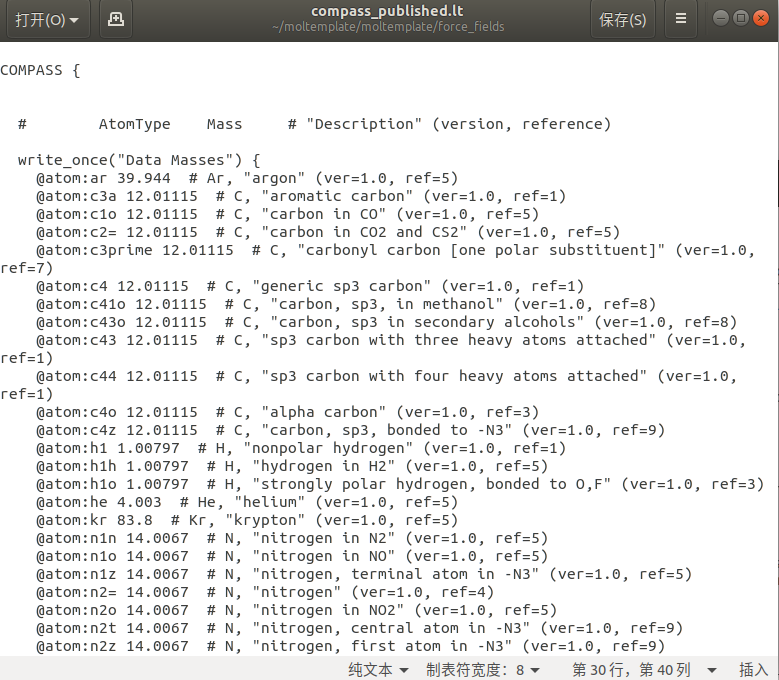
- moltemplate给出力场信息的方式是定义分子和系统的.lt文件,针对不同力场,moltemplate提供了对应力场的.lt文件,以Compass力场为例,moltemplate提供了compass_published.lt文件,其中定义了各种不同的原子,以及原子间成键的参数,成键类型,分子间作用力类型等等,在自己定义新的分子时,只需要继承compass_published.lt文件,然后指定分子中的原子种类及原子间的连接情况即可,moltemplate会自动为其分配质量、电荷、pair参数等信息,生成lammps可接受的输入文件.
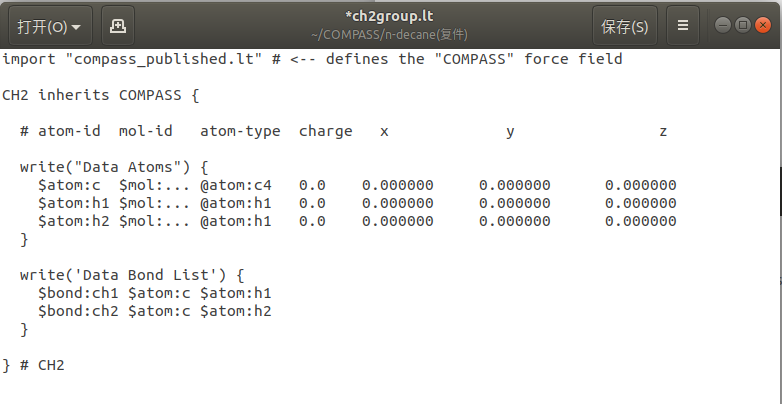
为了定义烷烃分子,可以先定义CH2基团和CH3基团,然后再把它拼起来组成分子,moltemplate也是基于这样的面向对象的想法设计的.下图定义了一个CH2分子,继承COMPASS力场,其中@atom是COMPASS力场文件中定义的原子类型,在这里就是c4和h1,charge可以随便写,之后moltemplate会根据力场文件重新生成. 后面的x y z是原子坐标,在完全用moltemplate生成结构文件时,必须通过计算得到.. 但是在这套技术栈之中我们就不需要指定坐标了,关键是指定这些原子的顺序,moltemplate完全按照最后系统中.lt文件中定义的各原子位置去给packmol生成的.pdb文件赋予力场信息.
ch2:
ch3:

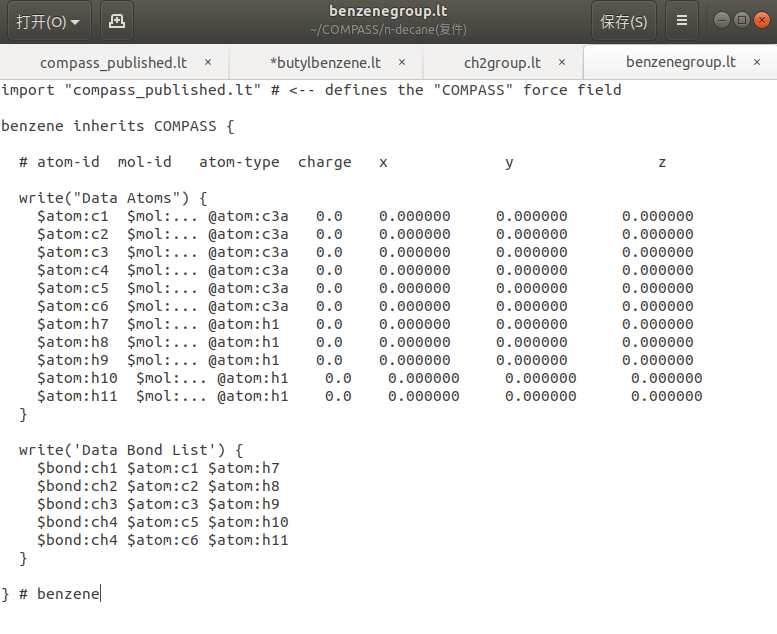
- 苯基:(完全按照pdb文件中的顺序,注意修改原子类型)
- 定义各分子的.lt文件(定义的顺序必须和gaussview生成的pdb文件中的原子顺序一致):
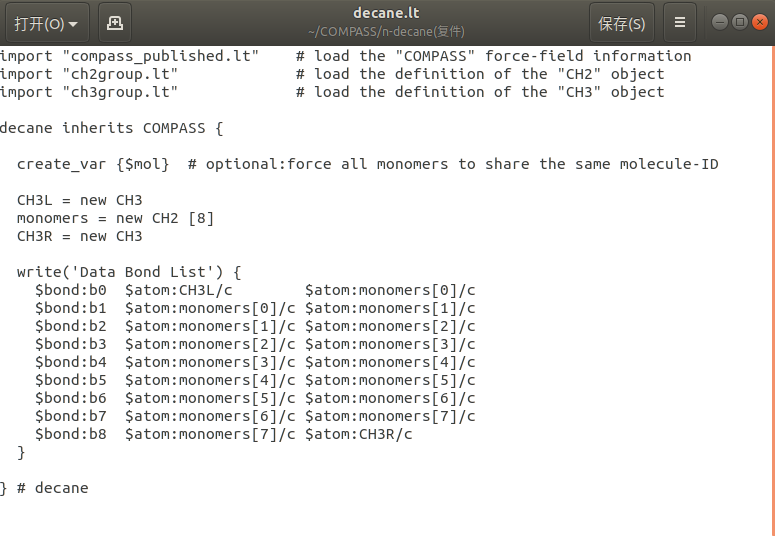
- decane:
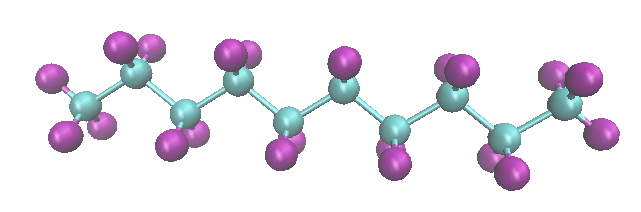
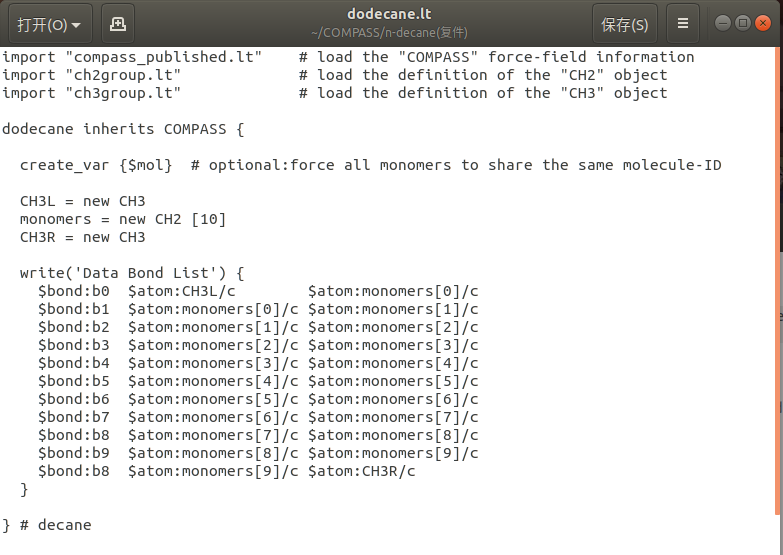
- dodecane:
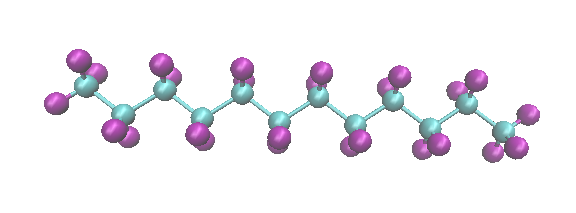
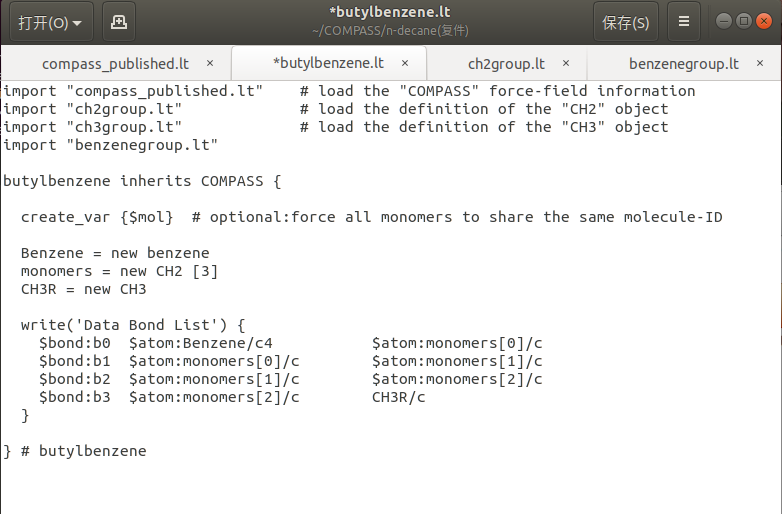
- butylbenzene:
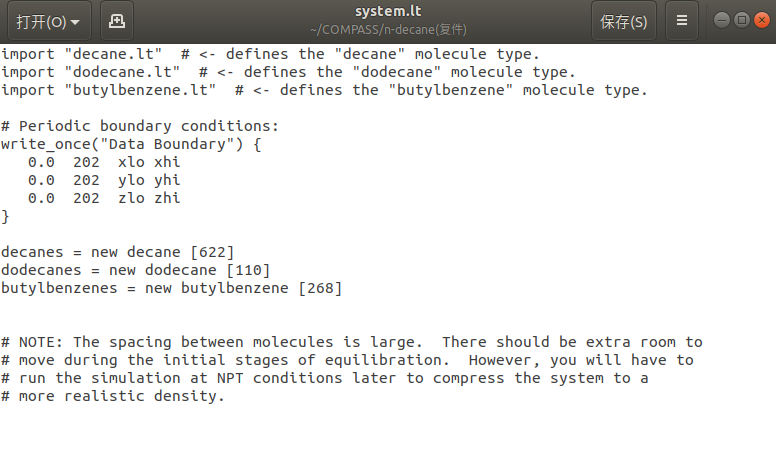
定义系统.lt文件,定义顺序必须和packmol空间排布定义的顺序完全一致.
运行:
1 | moltemplate.sh -pdb system.pdb system.lt |
用vmd看一下生成的system.data文件:

此时的data文件已经包含了compass力场所需的二面角等信息,moltemplate同时生成了settings和init等文件,进行lammps in文件的参数设置. 接下来只需要写in文件即可进行lammps模拟.